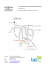A tutorial for cross-validation model with R
Transcription
A tutorial for cross-validation model with R
Université de Caen Basse-Normandie 1 11 septembre 2014 Département de Mathématiques et Mécanique Validation croisée avec R Sommaire 1.1 1.2 1.3 1.4 1.5 1.6 Objectifs . . . . . . . . . . . . Description . . . . . . . . . . Préparation des données . . Exploration . . . . . . . . . . Partitionnement . . . . . . . Modélisation . . . . . . . . . 1.6.1 Sélection de variables . . 1.6.2 Construction d’un modèle 1.6.3 Diagnostic d’un modèle . 1.7 Evaluation d’un modèle . . . 1.7.1 Matrice de confusion . . . 1.7.2 K Validation croisée . . . 1.8 Transfert du modèle . . . . . 1.1 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 1 1 2 2 3 4 4 4 5 5 5 5 6 Objectifs — Mise en place d’une étude de data-mining — Utilisation de R — Validation, validation croisée, K-validation croisée 1.2 Description Cette table concerne une étude sur une maladie du coeur. Plusieurs hôpitaux ont réalisé cette étude. — Cleveland Clinic Foundation [R. et al.(1989)R., A., W., Pfisterer, M., S., K., S., and V] — Hungarian Institute of Cardiology, Budapest — V.A. Medical Center, Long Beach, CA — University Hospital, Zurich, Switzerland Elles ont été étudiées en 89 par le laboratoire Center for machine learning and intelligent systems de l’université de californie Irvine. Un descriptif des bases de données peut être trouvée à https://archive.ics.uci.edu/ml/datasets/Heart+Disease. 1 2 3 nom age sex cp type quanti quali quali 4 trestbps quanti 5 chol quanti 6 fbs quali 7 restecg quali 8 9 thalach exang quanti quali 10 oldpeak quanti 11 slope quali 12 ca qualiordonne 13 thal quali 14 Y quali label modalités chest pain type resting blood pressure (in mm Hg on admission to the hospital) serum cholestoral in mg/dl (fasting blood sugar > 120 mg/dl) (1 = true ; 0 = false) resting electrocardiographic results maximum heart rate achieved exercise induced angina ST depression induced by exercise relative to rest the slope of the peak exercise ST segment number of major vessels (0-3) colored by flourosopy thal: 3 = normal ; 6 = fixed defect ; 7 = reversable defect http://www.math.unicaen.fr/~kauffman/cours female ;male asympt ;atyp angina ;non anginal ;typ angina f ;t left vent hyper ;normal ;st t wave abnormality no ;yes down ;flat ;up 0 ;1 ;2 ;3 fixed defect ;normal ;reversable defect normal ;malade 1 [email protected] Université de Caen Basse-Normandie 11 septembre 2014 Département de Mathématiques et Mécanique Table 1: Catalogue des variables Références [R. et al.(1989)R., A., W., Pfisterer, M., S., K., S., and V] Detrano R., Janosi A., Steinbrunn W., Pfisterer, Schmid J. M., Sandhu S., Guppy K., Lee S., and Froelicher V. International application of a new probability algorithm for the diagnosis of coronary artery disease. American Journal of Cardiology, 64:304–310, 1989. 1.2 Nous allons utiliser les données de l’hôpital de Cleveland. Elles sont disponibles au formats : arff http://www.math.unicaen.fr/~kauffman/data/heart-c.arff. csv http://www.math.unicaen.fr/~kauffman/data/heart-c.csv. Elle comporte 303 lignes et 14 colonnes. Dans R on peut les lire avec les instructions suivantes : DATA="http://www.math.unicaen.fr/~kauffman/data/" X=foreign::read.arff(paste(DATA,"heart-c.arff",sep="")) # 303x14 cleveland X=read.table(paste(DATA,"heart-c.csv",sep=""),sep=";",header=TRUE) # 303x14 cleveland 1.3 Préparation des données 1. Lire le fichier de données à l’aide de la fonction read.arff de la librairie foreign. 2. Transformer les modalités {” < 50”, ” > 50 1”} de la variable num en {′ normal′,′ malade′}. 3. Renommer la variable cible ( celle que l’on va modéliser) num en la variable Y. 4. Transformer la variable ca numérique en facteur ordonné grâce à la commande factor. DATA="http://www.math.unicaen.fr/~kauffman/data/" X=foreign::read.arff(paste(DATA,"heart-c.arff",sep="")) # 303x14 cleveland X$Y=factor(as.character(X$num), levels=c("<50",">50_1"), labels=c('normal','malade')) X$num=NULL X$ca=factor(X$ca,ordered=TRUE) # valeurs 0,1,2,3 1.4 Exploration 1. Trouver les variables ayant au moins une valeur manquante. variable ca thal effectif 5 2 Table 2 – Variables ayant au moins une valeur manquante 2. Construire un vecteur nommé X.quanti contenant les noms de toutes les variables quantitatives is.numeric, lapply. 3. Construire un vecteur nommé X.quali contenant les noms de toutes les variables qualitatives. setdiff 4. Pour chacune des variables quantitatives construire un diagramme de type boı̂tes à moustaches de cette variable en fonction de la variable cible. http://www.math.unicaen.fr/~kauffman/cours 2 [email protected] Université de Caen Basse-Normandie 11 septembre 2014 Département de Mathématiques et Mécanique 0 1 2 3 4 5 6 oldpeak normal malade Figure 1 – Boites à moustaches de oldpeak en fonction de Y 5. Pour chacune des variables qualitatives construire un diagramme de type mosaique : en absisse les modalités de cette variable, en ordonnée les pourcentages d’être malade contre celle d’être normale. 1.0 thal 0.0 0.2 0.4 0.6 0.8 malade normal fixed_defect normal reversable_defect Figure 2 – Y 1.5 Partitionnement Dans cette partie on partitionnne l’ensemble des lignes en 3 parties apprentissage Ce sont les lignes qui vont servir à identifier les individus test Ce sont des lignes qui vont servir à tester le modèle, elles sont utilsées par le data scientist dans l’étape de construction du modèle. http://www.math.unicaen.fr/~kauffman/cours 3 [email protected] Université de Caen Basse-Normandie 11 septembre 2014 Département de Mathématiques et Mécanique validation Ces lignes servent à l’évaluation finale du modèle, elles n’ont absolument pas été utilisées dans les étapes d’apprentisssage ou de validation du modèle. Créer une variable nommée partition définissant les parties apprentissages, test et validation ( 60 %, 20%, 20%) de la base de données. classes=sample(1:3,nrow(X),replace=TRUE,prob=c(0.6,0.2,0.2)) X$partition=factor(classes, levels=1:3, labels=c('apprentissage','test','validation')) table(X$partition)/nrow(X) 1.6 1.6.1 Modélisation Sélection de variables 1. A l’aide la commande graphique suivante plot(Y~.,data=X) sélectionner des variables pouvant influencer la variable cible. 2. On choisi les variables suivantes variables=c('thal','ca','cp') formule=Y~thal+ca+cp plot(formule,data=X) On va expliquer la variable cible uniquement en fonction des variables sélectionnées thal, ca, cp. 1.6.2 Construction d’un modèle Dans ce paragraphenon construit un arbre de décision expliquant la variable qualitative binaire Y en fonction des trois variables sélectionnées. library(rpart) arbre=rpart(formule,data=X[X$partition=="apprentissage",],method="class") plot(arbre) text(arbre) post(arbre,file="ps/R-arbre.eps")#$ sortie postscript print(arbre) # affichage texte Figure 3 – Arbre de décision > arbre n= 212 http://www.math.unicaen.fr/~kauffman/cours 4 [email protected] Université de Caen Basse-Normandie 11 septembre 2014 Département de Mathématiques et Mécanique node), split, n, loss, yval, (yprob) * denotes terminal node 1) root 212 96 normal (0.54716981 0.45283019) 2) thal=normal 117 26 normal (0.77777778 0.22222222) 4) ca=0,1 100 14 normal (0.86000000 0.14000000) 8) trestbps< 152 89 8 normal (0.91011236 0.08988764) 16) slope=down,up 54 1 normal (0.98148148 0.01851852) * 17) slope=flat 35 7 normal (0.80000000 0.20000000) 34) age< 53.5 15 0 normal (1.00000000 0.00000000) * 35) age>=53.5 20 7 normal (0.65000000 0.35000000) 70) age>=63.5 10 0 normal (1.00000000 0.00000000) * 71) age< 63.5 10 3 malade (0.30000000 0.70000000) * 9) trestbps>=152 11 5 malade (0.45454545 0.54545455) * 5) ca=2,3 17 5 malade (0.29411765 0.70588235) * 3) thal=fixed_defect,reversable_defect 95 25 malade (0.26315789 0.73684211) 6) ca=0 42 21 normal (0.50000000 0.50000000) 12) exang=no 20 2 normal (0.90000000 0.10000000) * 13) exang=yes 22 3 malade (0.13636364 0.86363636) * 7) ca=1,2,3 53 4 malade (0.07547170 0.92452830) * 1.6.3 1.7 1.7.1 Diagnostic d’un modèle Evaluation d’un modèle Matrice de confusion Pour chacun des individus, on va calculer la prédiction du modèle malade ou normal. On va construire la matrice de confusion qui consiste à énumérer les différentes possibilités d’une personne d’être dans un des groupe malade, normal et d’être prédit par le modèle dans un des groupes malade, normal. normal malade normal estime 76 12 malade estime 21 67 Table 3 – Matrice de confusion sur la base d’appentissage Une possibilité pour évaluer ce modèle est de calculer sa proportion d’erreurs de prédiction : 21+12 76+21+12+67 ∼ 19% X$Y.chap=predict(arbre,newdata=X,type="class") # erreur sur base d'apprentissage subset=X$partition=="apprentissage" res=table(X$Y[subset],X$Y.chap[subset]) (erreur=1-sum(diag(res))/sum(res)) 1. Calculer la matrice de confusion sur la base de test 2. Calculer le taux d’erreur sur la base de test. 3. Quelle erreur faut-il choisir calculée sur la base de test ou la base d’apprentissage, discuter ? 1.7.2 K Validation croisée La K-validation croisée consiste 1. à partitionner les individus en K parties d’effectifs approximativement égal. 2. Pour chacune de ce ces K parties (a) On identifie le modèle sur toutes les autres K − 1 parties (b) On évalue alors le modèle sur la partie sélectionnée avec le modèle calculé à l’aide des K − 1 autres parties. 3. On peut alors étudier les évaluations de K modèles, moyenne, écart type, coeffcient de variation. Ces évaluations sont plus réalistes : c’est à dire pas trop optimistes et reflètent la variabilité théorique de l’estimateur de l’évaluation. http://www.math.unicaen.fr/~kauffman/cours 5 [email protected] Université de Caen Basse-Normandie 11 septembre 2014 Département de Mathématiques et Mécanique Pour déterminer une partition en K parties d’effectif approximativement égal, on distingue deux types de méthodes. approximative On tire au hasard avec remise parmi K entiers de façon équiprobable les parties sont donc d’effectifs approximativent égales. exacte On tire au hasard n échantillons d’une variable aléatoire, chacun de ces nombres est affecté à un individu, on calcule alors les quantiles (qi )i=1..,K−1 d’ordre 1/K, 2/K, · · · , (K − 1)/K . Les parties sont alors déterminées par les intervalles ] − ∞, q1 ], ]q1 , q2 ], · · · , ]qK−1 , +∞[. 1. Pour chaque individu (ligne) tirer au hasard avec remise parmi {1, · · · , K} de façon équiprobable. La classe affectée sera le numéro tiré. k=10 n=nrow(X) X$classe=factor(sample(1:k,nrow(X),replace=TRUE,prob=rep(1/k,k))) > table(X$classe) 1 2 3 4 5 6 7 8 9 10 37 23 24 34 30 32 23 43 31 26 2. C’est la méthode pour avoir des parties ayant des effectifs presque égaux. taille=nrow(X) %/% k alea=runif(n) X$classe=cut( alea, breaks=c("0%"=0, quantile(alea,probs=seq(from=1/k,to=1-1/k,by=1/k)), "100%"=1),labels=FALSE) > table(X$classe) # la keme classe n'a pas de nom 1 2 3 4 5 6 7 8 9 10 31 30 30 30 31 30 30 30 30 31 3. Dans cette question, on réalise la méthode de K validation croisée. erreurs=c() for ( fold in 1:k ) { training=X$classe!=fold test=X$classe==fold arbre=rpart(Y~.,data=X[training,c(variables,"Y")],method="class") resultat=table(predict(arbre,newdata=X[test,c(variables,"Y")],type="class"), X$Y[test]) erreurs=c(erreurs,1-sum(diag(resultat))/sum(resultat)) } #$ > sum(erreurs*table(X$classe))/sum(table(X$classe)) [1] 0.1617162 > mean(erreurs) # classe m^ eme effectif [1] 0.1619355 > sd(erreurs) [1] 0.03401885 > (cv=sd(erreurs)/mean(erreurs)) [1] 0.2100766 1.8 Transfert du modèle Il s’agit maintenant de réaliser le modèle en Java, Excel, pour que le client final puisse prédire la variable cible sur une nouvelle base de données. http://www.math.unicaen.fr/~kauffman/cours 6 [email protected] Université de Caen Basse-Normandie http://www.math.unicaen.fr/~kauffman/cours 11 septembre 2014 7 Département de Mathématiques et Mécanique [email protected]